環境DNA解析
環境DNA解析とは
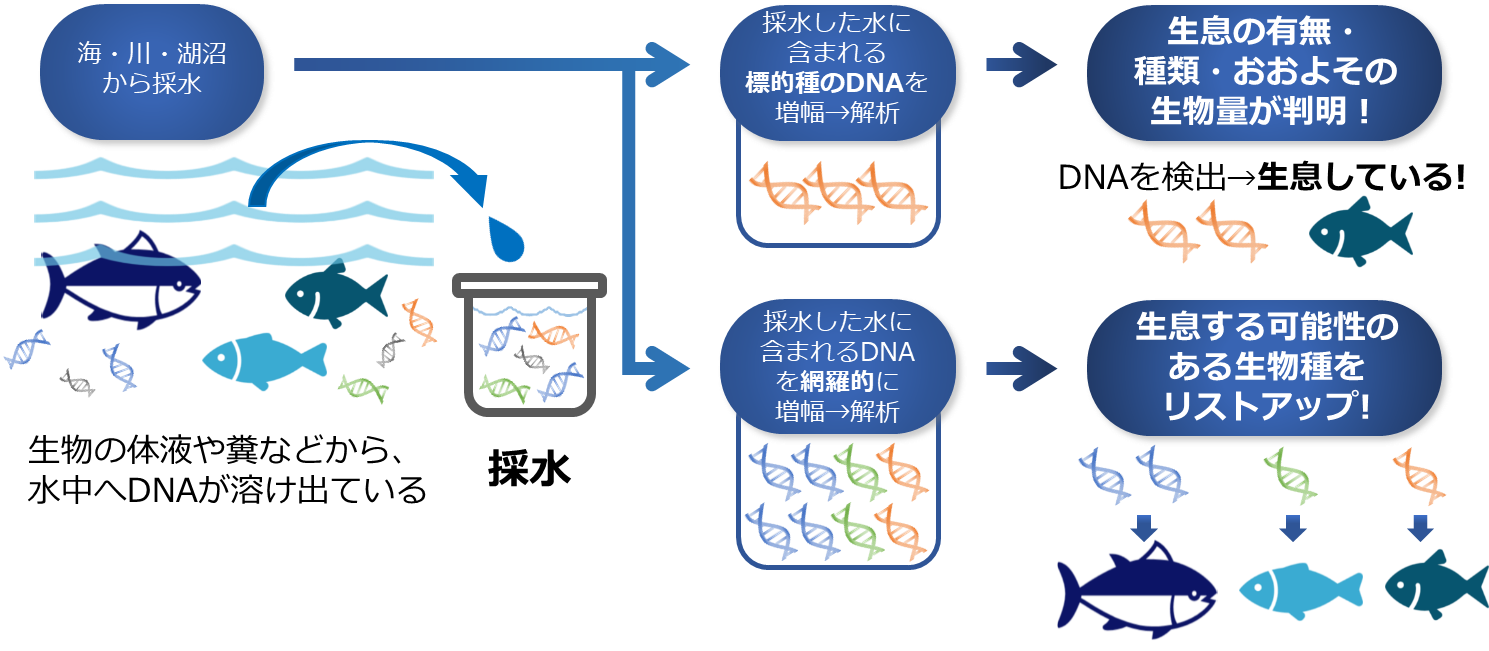
- 環境DNAとは、海や川・湖沼・土壌などの環境 中に存在する生物由来のDNAを指します。
- 環境DNAを解析することで、そこに生息する生物の種類やおおよその生物量の把握が可能です。
- 環境DNAを用いると調査が採水のみで可能となるため、これまで調査にかかっていた労力や環境への負荷などを削減できる画期的な調査手法として注目されています。
環境DNA解析を生物調査に適用するメリット
- 対象種を捕獲する必要が無く、調査地を荒らさずに対象種の生息確認が出来ます!
- 調査地では採水のみなので、短時間に広い範囲を調査可能です!
- 捕獲が困難な種(絶滅危惧種や導入初期の外来生物)の分布把握が可能です!
- 水産資源量の推定にも応用が可能です!

環境DNA解析の基本サービス
- 特定の生物種がいるかを知りたい!
→ Real time PCR - 特定の生物種のDNA量をはかりたい!
→ 定量PCR(qPCR)
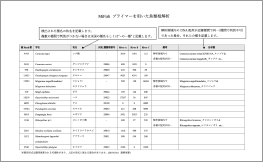
- 対象種のDNAのみを増幅させるプライマーを設計します
- 定量PCRの場合はプローブも設計します
※ポジティブコントロールとして肉片もしくは確実に生息していた水サンプルを入手できることが望ましいです
- 調査地点の生物の種組成を知りたい!
→ メタバーコーディング - 複数地点で多種の量的な比較がしたい!
→ 定量メタバーコーデイング
(Tourlousse et al.2017, Ushio et al.2018)
 魚類:MiFish-U, U2, E ※2,※3
魚類:MiFish-U, U2, E ※2,※3 哺乳類:MiMammal
哺乳類:MiMammal 甲殻類:MiDeca
甲殻類:MiDeca 鳥類:MiBird
鳥類:MiBird 細菌:16S rRNA遺伝子(V3-V4領域)
細菌:16S rRNA遺伝子(V3-V4領域)
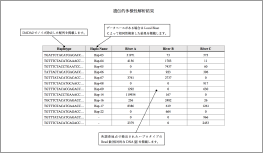
既知濃度の合成DNAをサンプルに混ぜ合わせる事で、同時に多種の定量が可能&PCR阻害も考慮した評価が可能!
※2 Ev2, U2はUでは増幅されにくい分類群のDNAを増幅させるプライマー
※3 Ev2, U2以外の追加プライマーでの対応も可能です
- 同種内の遺伝子の多様性を解読したい!
→ Miseqによる塩基配列決定
→ DADA2によるデノイス
(Callahan et al.2016)
- Sigsgaard et al. 2018 : イルカの遺伝的多様性
- Parsons et al.2018:ネズミイルカの遺伝的多様性
- Tsuji et al.2019:アユの遺伝的多様性
一つのサンプルで複数の検出方法・検出種を組み合わせることも可能です!
※価格はお問い合わせ下さい。
※定量分析を実施する場合には、追加費用が必要です。
- MiFish pipelineによる魚類の分子同定
- 遺伝的多様性解析:DADA2、生物種の分子同定:Usearch
種の特定のみならず、群集構造解析も提供することが可能です!
お気軽にお問い合わせください!
過去の検出事例
| 対象生物 | |
|---|---|
| Fish | 魚類 |
| Hynobius Andrias Onychodactylus |
サンショウウオ属 オオサンショウウオ属 ハコネサンショウウオ属 |
| Frog | カエル類 |
| Mammal | 哺乳類 |
| Avian | 鳥類 |
| Algae | 藻類 |
| Eukaryota | 真核生物 |
| Bacteria | 細菌叢 |
| Prokaryota | 原核生物 |
| Ammonia oxidizing bacteria | アンモニア酸化細菌 |
| Water invertebrate Insecta |
水生昆虫 |
| Decapoda | 甲殻類 |
| 対象生物 | |
|---|---|
| Liobagrus reini | アカザ |
| Cynops pyrrhogaster | アカハライモリ |
| Plecoglossus altivelis | アユ |
| Mauremys japonica | イシガメ |
| Acheilognathus longipinnis | イタセンパラ |
| Parahucho perryi | イトウ (核DNA) |
| Parahucho perryi | イトウ (ミトコンドリアDNA) |
| Salvelinus sp. | イワナ属 |
| Lithobates catesbeianus | ウシガエル |
| Micropterus salmoides salmoides | オオクチバス |
| Andrias japonicus | オオサンショウオ |
| Engraulis japonicus | カタクチイワシ |
| Chimarrogale platycephalus | カワネズミ |
| Limnoperna fortunei | カワヒバリガイ |
| Oryzias sakaizumii | キタノメダカ |
| Cyprinus carpio | コイ |
| Micropterus dolomieu | コクチバス |
| Oncorhynchus masou (masou, isikawae) | サクラマス・サツキマス |
| Oncorhynchus keta | シロサケ |
| Anguilla japonica | ニホンウナギ |
| Cambaroides japonicus | ニホンザリガニ |
| Hynobius hidamontanus | ハクバサンショウウオ |
| Onychodactylus japonicus | ハコネサンショウウオ |
| Opsariichthys uncirostris | ハス |
| Actinobacteria | 放線菌 |
| Lefua echigonia | ホトケドジョウ |
| Acheilognathus tabira jordani | ミナミアカヒレタビラ (富山) |
| Oryzias latipes | ミナミメダカ |
| 対象生物 | |
|---|---|
| Plecoglossus altivelis altivelis | アユ |
| Salvelinus | イワナ |
| 対象生物 | |
|---|---|
| Mustela sp. | イタチ (ニホンイタチ ⇔ チョウセンイタチ) |
| Lethenteron sp. | スナヤツメ (南方種 ⇔ 北方種) |
| Rana sp. | タゴガエル (ネバタゴガエル ⇔タゴガエル) |
| Martes sp. | テン (テン ⇔ クロテン) |
| Misgurnus sp. | ドジョウ (キタドジョウ ⇔ ドジョウ) |
| - | 哺乳類の糞便のホスト種 |
目に見えない生物も・・・
16S rRNA遺伝子(V3-V4領域)をPCRで増幅することで、環境中の細菌叢解析を行います。
ご要望に応じて既報の特異的な検出系での分析や、新たな検出系の設計も行います。
また、細菌以外の微生物に対しての菌叢解析もご相談に応じて対応いたします。
上記以外についてもぜひお問合せください
解析
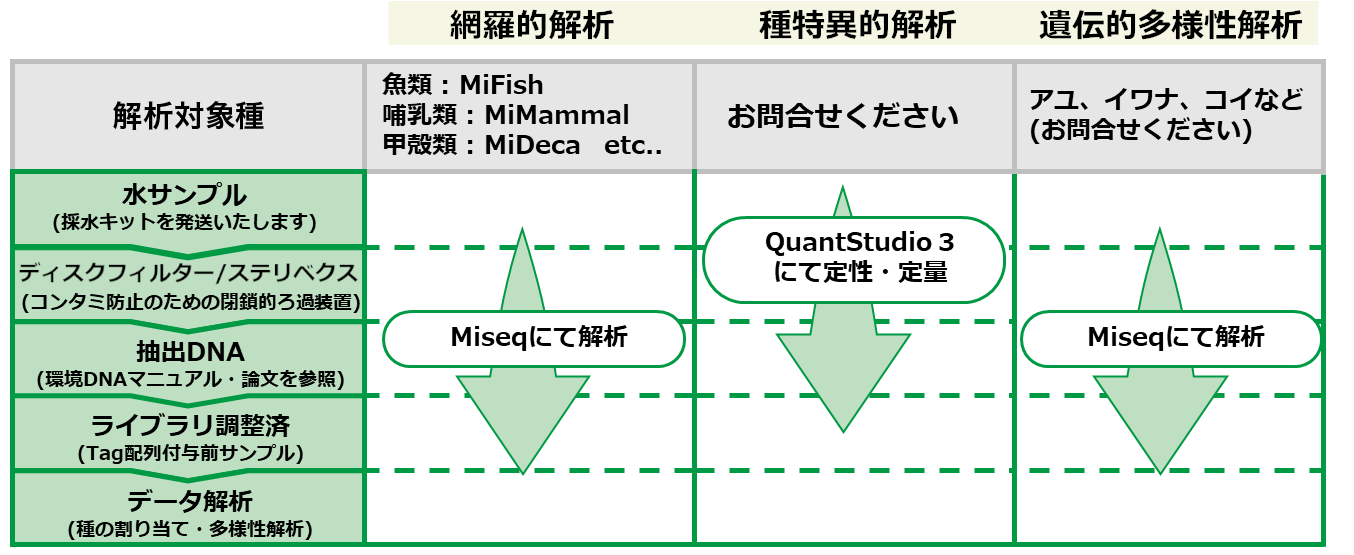
網羅的解析は主に、魚類、哺乳類、鳥類、細菌叢を対象としています。
MiFishを用いた魚類相解析でweb解析をご希望の場合は公式のチュートリアルに従い、原則デフォルト設定で解析を実施いたします。また、設定値のご希望がある場合はメールまたは分析依頼書の備考欄に記載いただきますようお願いいたします。
また、弊社ではお客様へのご質問対応と検出精度向上のために、魚類相はMiya et al.2015, 哺乳類相,鳥類相はUshio et al.2017, 2018に従いデータベースを作成、Takeuchi et al.2019, Komai et al.2019に従いパイプラインを作成し、社内で解析を実施しております。
遺伝的多様性解析は1塩基多型を識別する必要があることからTsuji et al.2019に従いDADA2 (Callahan et al.2016)を用いた解析を実施しております。
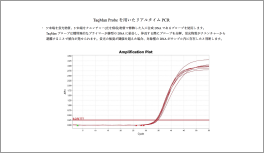
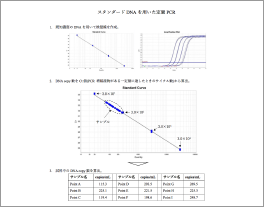
種特異的解析はQuantStudio 3を用いて、リアルタイムPCRによる検出を実施しております。 また、定量を行う場合には、確実に検出されるサンプルが必要なため、対象種の組織片もしくは飼育水などの入手が必要になる場合があります。
引用文献
- Callahan, Benjamin J., et al. "DADA2: high-resolution sample inference from Illumina amplicon data." Nature methods 13.7 (2016): 581.
- Komai, Tomoyuki, et al. "Development of a new set of PCR primers for eDNA metabarcoding decapod crustaceans." Metabarcoding and Metagenomics 3 (2019): e33835.
- Miya, Masaki, et al. "MiFish, a set of universal PCR primers for metabarcoding environmental DNA from fishes: detection of more than 230 subtropical marine species." Royal Society open science 2.7 (2015): 150088.
- Parsons, Kim M., et al. "Water, water everywhere: environmental DNA can unlock population structure in elusive marine species." Royal Society open science 5.8 (2018): 180537.
- Sato, Yukuto, et al. "MitoFish and MiFish pipeline: a mitochondrial genome database of fish with an analysis pipeline for environmental DNA metabarcoding." Molecular biology and evolution 35.6 (2018): 1553-1555.
- Sigsgaard, Eva Egelyng, et al. "Population characteristics of a large whale shark aggregation inferred from seawater environmental DNA." Nature ecology & evolution 1.1 (2017): 0004.
- Takeuchi, Aya, et al. "New PCR primers for metabarcoding environmental DNA from freshwater eels, genus Anguilla." Scientific reports 9.1 (2019): 7977.
- Tourlousse, Dieter M., Akiko Ohashi, and Yuji Sekiguchi. "Sample tracking in microbiome community profiling assays using synthetic 16S rRNA gene spike-in controls." Scientific reports 8.1 (2018): 9095.
- Tsuji, Satsuki, et al. "The detection of aquatic macroorganisms using environmental DNA analysis—A review of methods for collection, extraction, and detection." Environmental DNA (2019).
- Tsuji, Satsuki, et al. "Evaluating intraspecific genetic diversity using environmental DNA and denoising approach: A case study using tank water. Environmental DNA." Environmental DNA
- Ushio, Masayuki, et al. "Demonstration of the potential of environmental DNA as a tool for the detection of avian species." Scientific reports 8.1 (2018): 4493.
- Ushio, Masayuki, et al. "Environmental DNA enables detection of terrestrial mammals from forest pond water." Molecular Ecology Resources 17.6 (2017): e63-e75.
- Ushio, Masayuki, et al. "Quantitative monitoring of multispecies fish environmental DNA using high-throughput sequencing." Metabarcoding and Metagenomics 2 (2018): e23297.